Projekte
Nachweis der geografischen Herkunft von Erdbeeren

Die Problematik der Herkunftsverfälschung auf dem Erdbeermarkt ist schon seit längerer Zeit bekannt, da zunehmend beobachtet wird, dass deutsche Erdbeeren mit ausländischer Ware gestreckt werden. Aufgrund der großen Preisunterschiede bietet eine Umdeklaration einen hohen finanziellen Anreiz, allerdings werden dadurch neben den Verbrauchern massiv die redlichen deutschen Erdbeeranbauer geschädigt.
Derzeit stehen der Erdbeerbranche keine analytischen Methoden zur Verfügung, die einen verlässlichen Nachweis der Herkunft ermöglichen. Die üblicherweise angewendete Stabilisotopenanalyse liefert nicht immer eindeutige Ergebnisse und kann nur von sehr spezialisierten Forschungseinrichtungen durchgeführt werden, sodass kein flächendeckender Einsatz möglich ist.
Um sowohl die redlichen Erdbeerbauern als auch die Verbraucher vor Betrug zu schützen, sollen alternative analytische Methoden zur Bestimmung der Herkunft entwickelt werden, die auf einer objektiven Datenerfassung beruhen. Zum Einsatz sollen zwei komplementäre Ansätze (LC-MS/MS und ICP-MS) kommen, da bislang nicht absehbar ist, welche Technologie für einen Herkunftsnachweis besser geeignet ist. In jedem Fall liefert die chemometrische Zusammenführung (data fusion) der unterschiedlichen Datensätze ein höher aufgelöstes chemisches Abbild der jeweiligen Erdbeeren unterschiedlicher Herkünfte, was gerade bei Gebieten, die bspw. an der Grenze zu Nachbarländern liegen, von unschätzbarem Wert sein kann.
Nach dem erfolgreichen Abschluss des Projekts können die entwickelten Methoden unmittelbar in Qualitätssicherungslaboratorien der KMU, in Handelslaboratorien sowie in den Laboratorien der Landesuntersuchungsämter eingeführt werden. Das Projekt ist darauf abgestimmt, dass die Umsetzung mit der typischen Laborausstattung erzielt werden kann bzw. nur geringfügige Zusatzinvestitionen getätigt werden müssen. Alternativ besteht für die Unternehmen die Möglichkeit, die Analysen an Auftragslabore zu vergeben.
Dieses Projekt wird von der im Programm zur Förderung der “Industriellen Gemeinschaftsforschung (IGF)“ vom Bundesministerium für Wirtschaft und Klimaschutz (via AiF) über den Forschungskreis der Ernährungsindustrie e.V. (FEI) unter der Projektnummer AiF 22909 N gefördert und von Kim Brettschneider in Kooperation mit der Arbeitsgruppe von Markus Fischer bearbeitet.
Authentifizierung von Honig

Omiks-Technologien werden heutzutage immer wichtiger, um verschiedene biologische Proben, wie zum Beispiel Lebensmittel, zu charakterisieren und zu klassifizieren. Im Gegensatz zu Genomics und Transkripomics wird die Metabolomics Ebene stark durch die Umwelt der untersuchten Probe beeinflusst. Insbesondere das Metabolom von Honigerzeugnissen ist von Umwelteinflüssen geprägt, da Honig von Bienen produziert wird, die Nektar und Pollen aus ihrer unmittelbaren Umgebung sammeln. Um das Metabolom biologischer Proben zu analysieren, ist das analytische Verfahren der Flüssigchromatographie mit Massenspektrometrie-Kopplung (LCMS) aufgrund ihres hohen Durchsatzes, der optionalen weichen Ionisierung und der breiten Erfassung bestimmter Verbindungsklassen sehr gut geeignet. Das Ziel dieses Projektes ist es, die Authentizitätanalyse von Honig mit einer geeigneten bioinformatischen Analyse der LCMS Daten zu ermöglichen.
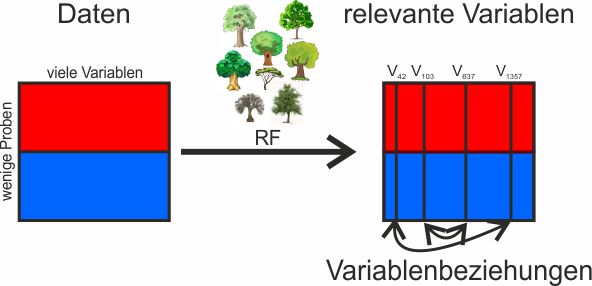
Dieses Projekt ist in drei Teile gegliedert. Im ersten Teil wird ein Workflow entwickelt, durch den LCMS-Daten von verschiedenen Geräten genutzt werden können. Anschließend werden chemometrische Methoden wie die Hauptkomponentenanalyse (PCA), Soft Independent Modelling of Class Analogy (SIMCA) und Methoden des maschinellen Lernens, z. B. Random Forest (RF), zur Unterscheidung von Honigproben angewendet. Neben der Anwendung von RFs zur Klassifizierung werden RF-basierte Verfahren dabei auch zur Charakterisierung des Honigs verwendet. Dies wird dadurch erreicht, dass relevante Variablen selektiert werden, welche für die Unterschiede zwischen den Proben relevant sind und damit für die Interpretation spezifischer Umwelteinflüsse verwendet werden können. Darüber hinaus werden die Variablenbeziehungen der komplexen Daten mit Surrogate Minimal Depth (SMD) untersucht. SMD ermöglicht dabei die Identifizierung von gemeinsam vorkommenden Metaboliten und metabolischen Pathways, die mit bestimmten Umwelteinflüssen und somit mit der geografischen Herkunft des Honigs in Verbindung gebracht werden können.
Im zweiten Teil dieses Projekts wird die Verfälschung von Honig durch Zuckersirupe analysiert. Dazu werden multivariate Regressionsmodelle, z.B. durch Partial Least Squares (PLS) Regression oder RF erstellt, um den Anteil des dem Honig zugesetzten Sirups zu bestimmen. Auch hier werden Variablenselektionsverfahren eingesetzt, um spezifische Marker für Zuckersirupe zu identifizieren.
Die in den ersten beiden Teilen des Projekts entwickelten Methoden werden anschließend im dritten Teil des Projekts auf andere Bereiche der Lebensmittelauthentifizierung, wie z. B. die Verfälschung von Fruchtsäften, angewendet.
Dieses Projekt wird von Jule Hansen bearbeitet.
Charakterisierung von Lebensmitteln anhand ihres Metaboloms
Die umfassende Analyse des Metaboloms wird für die Charakterisierung und Klassifizierung verschiedener biologischer Proben, einschließlich Lebensmitteln, immer wichtiger. Das Metabolom umfasst dabei mehrere sehr unterschiedliche Molekülklassen, weshalb verschiedene Analysetechniken für die Metabolomanalyse entwickelt wurden. Die meisten von ihnen basieren auf NMR und Massenspektrometrie. Bei der Lebensmittelanalyse wird Metabolomics zur Aufdeckung von Betrug eingesetzt, z.B. durch die Bestimmung der geografischen Herkunft und taxonomischer Merkmale.
In diesem Projekt werden bioinformatische Verfahren für die umfassende Analyse verschiedener Metabolomics-Daten und damit für die Klassifizierung, Charakterisierung und Authentifizierung von Lebensmitteln entwickelt. Dies wird durch die Anwendung etablierter Methoden wie der Hauptkomponentenanalyse (PCA) und Methoden des maschinellen Lernens, z. B. Random Forest (RF), erreicht. Letztere bieten verschiedene Vorteile für die Analyse von Metabolomics-Daten: Neben der Anwendung von RF zur Klassifizierung werden RF-basierte Ansätze auch zur Charakterisierung der untersuchten Lebensmittelproben eingesetzt. Dies geschieht durch die Selektion relevanter Variablen, die zur Interpretation von Unterschieden zwischen den Klassen und damit von spezifischen Einflüssen auf das Metabolom von Lebensmitteln verwendet werden. In diesem Zusammenhang werden verschiedene Selektionsverfahren angewendet und verglichen. Darüber hinaus werden die komplexen Eigenschaften des Metaboloms durch die Analyse von Variablenbeziehungen erfasst. Diese wird durch die Anwendung von Surrogate Minimal Depth (SMD) erreicht und ermöglicht die Identifizierung von gemeinsam vorkommenden Metaboliten, bzw. metabolischer Pathways, die mit spezifischen Umwelteinflüssen von Lebensmitteln in Verbindung gebracht werden können. Um dieses Wissen über funktionelle Beziehungen anschließend direkt in den Modellierungsprozess einzubeziehen, werden in diesem Projekt auch sog. pathway-guided RF Methoden angewendet.
Dieses Projekt wird von Sören Wenck bearbeitet.
Authentifizierung von Lebensmitteln mit mobiler NIR-Spektroskopie

In der globalen Rohstoff- und Lebensmittelversorgungskette gewinnen schnelle und zuverlässige Verfahren zur Qualitäts- und Eingangskontrolle zunehmend an Bedeutung. Besonders gefragt sind Untersuchungsmethoden, die nicht nur in kurzer Zeit präzise Ergebnisse liefern, sondern auch preiswert, leicht anwendbar und für den Einsatz mit minimaler technischer Ausrüstung geeignet sind. In den letzten Jahren sind einige Handheld Geräte entwickelt worden, die diese Kriterien durch die Anwendung der Nahinfrarot(NIR)-Spektroskopie erfüllen. Dabei werden NIR-Spektren erhalten, die durch überlagernde Signale einer Vielzahl verschiedener Moleküle charakterisiert sind und als Fingerabdruck der Zusammensetzung der untersuchten Probe mit multivariaten Methoden ausgewertet werden.
Das Ziel dieses Projekts besteht darin, diesen Ansatz für die Untersuchung verschiedener Lebensmittel anzuwenden, um schnelle und einfache Authentifizierungsverfahren zu entwickeln. Dazu wird eine breite Palette unterschiedlicher Lebensmittel untersucht. Die bereits begonnene Analyse von Nüssen, also fett- und proteinreicher pflanzlicher Lebensmittel, soll dabei vertieft und auf die Analyse fett- und proteinreicher tierischer Lebensmittel wie Fisch ausgedehnt werden. Dabei soll u.a. analysiert werden, ob Unterschiede bei der Fermentierung von Matjes detektiert werden können. Dies ist deshalb relevant, da die Fermentation zeit- und kostenintensiv, aber auch für die Qualität des Matjes entscheidend ist. Eine Herausforderung dabei könnte, wie es bei der Anwendung der NIR-Spektroskopie auf wasserreiche Lebensmitte häufig der Fall ist, die Überlagerung relevanter spektraler Informationen durch intensive Wasserbanden sein. Eine Lebensmittelklasse bei der dieses Problem nicht auftritt sind Gewürze, da diese meist in getrockneter Form verkauft werden. Fragestellungen, die in diesem Zusammenhang untersucht werden, sind die Detektion von Kurkumabeimischungen in gemahlenem Safran und die Streckung von Ceylon Zimt mit Cassia Zimt.
Da, wie zuvor beschrieben, NIR-Spektren als Fingerabdruck der biologischen Proben verwendet werden, besteht ein weiteres Ziel des Forschungsprojekts darin, das Verständnis der erzeugten Spektren im Kontext der Lebensmitteluntersuchung zu vertiefen. Durch die Fusionierung der NIR Daten mit massenspektrometrischen und kernspinresonanzspektroskopischen Daten soll ein tieferes Verständnis darüber gewonnen werden, welche Informationen die mobilen NIR-Geräte erfassen und wie diese zur Klassifizierung der Lebensmittelproben beitragen.
Dieses Projekt wird von Nicolas Lauer bearbeitet.
Analyse von Schriftartefakten
Dieses Projekt ist Teil des Exzellenzclusters "Understanding Written Artefacts" und wird von Lucas Voges bearbeitet. Weitere Informationen zum Projekt sind hier zu finden.
Oberflächenverstärkte Raman-Streuung
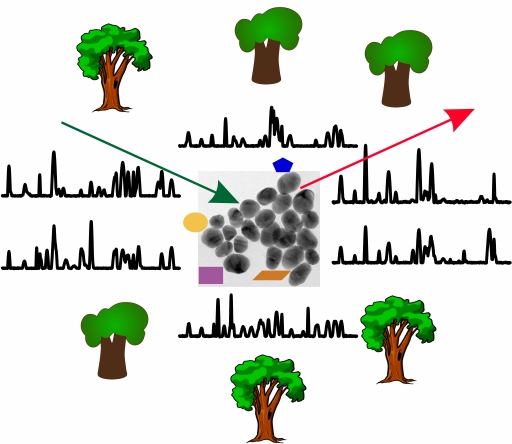
Die oberflächenverstärkte Raman-Streuung (engl. surface-enhanced Raman scattering, SERS) in Abwesenheit spezifischer Labels oder Reportermoleküle wird vermehrt zur Charakterisierung der Zusammensetzung und Struktur von biologischen Proben eingesetzt. SERS basiert auf der Wechselwirkung von Molekülen bzw. spezifischen funktionellen Gruppen mit Metallnanostrukturen und macht diese sichtbar. Aufgrund der variierenden Interaktionen zwischen Molekülen und Metallnanopartikeln entstehen hochkomplexe Daten, die einer anspruchsvollen Auswertung bedürfen, wenn sie der Charakterisierung von Struktur bzw. Molekül-Metall- oder Molekül-Molekül-Wechselwirkung dienen sollen. Dies ist besonders dann der Fall, wenn Proben untersucht werden, die aus unterschiedlichen molekularen Spezies bestehen und durch verschiedene Bedingungen, wie z.B. den pH-Wert oder relative Konzentrationen, beeinflusst werden. Um SERS Daten überhaupt nutzbar zu machen, ist es deshalb notwendig, leistungsfähige Auswertungsverfahren zu entwickeln.
In diesem Projekt werden Random Forest (RF) basierte Methoden, die sich in ersten Anwendungen auf reale SERS-Daten als sehr robust erwiesen haben, für die Analyse von Daten aus Modellversuchen konzeptuell erprobt und angepasst. Die Daten werden unter genau definierten experimentellen Bedingungen erzeugt. Dabei wird die Komplexität der hier verwendeten Systeme schrittweise von einzelnen molekularen Komponenten wie Lipidmembranen oder Wirkstoffmolekülen, über die Kombination zweier Komponenten bis zur komplexen Umgebung endolysosomaler Vesikel in Zellen erhöht. Die RF-Analyse dient dabei der Erreichung der drei folgenden Ziele: (i) der Selektion spektraler Merkmale (Variablen) für eine direkte Strukturinterpretation, (ii) der Identifizierung gleichzeitig auftretender spektraler Merkmale, damit die Wechselwirkung verschiedener Moleküle beobachtet werden kann und (iii) der Integration von a priori Wissen. Hierzu gehören insbesondere SERS-Spektren aus jeweils anderen Experimenten in diesem Projekt. Die Nutzbarmachung der RF-Analyse-Modelle beruht auf dem unterschiedlichen Komplexitätsgrad der SERS-Experimente sowie auf einer systematischen Wahl und Modifizierung der experimentellen Bedingungen. Zusätzlich zu den experimentell generierten Daten werden im Laufe des Projekts auch SERS-Daten simuliert, um die Auswirkungen bestimmter Umgebungsbedingungen gezielt einzubeziehen und die Ergebnisse iterativ mit denen der Experimente zu vergleichen. Die Ergebnisse dieses Projekts sind ein wichtiger Schritt hin zum Einsatz von SERS für die Charakterisierung von biophysikalischen Modellen und von Molekülen in komplexen biologischen Umgebungen.
Dieses Projekt wird von der DFG unter der Projektnummer 511107129 gefördert und von Florian Gärber in Kooperation mit der Arbeitsgruppe von Janina Kneipp an der Humboldt-Universität Berlin bearbeitet.
