Abgeschlossene Projekte
Authentifizierung von Honig

Omiks-Technologien werden heutzutage immer wichtiger, um verschiedene biologische Proben, wie zum Beispiel Lebensmittel, zu charakterisieren und zu klassifizieren. Im Gegensatz zu Genomics und Transkripomics wird die Metabolomics Ebene stark durch die Umwelt der untersuchten Probe beeinflusst. Insbesondere das Metabolom von Honigerzeugnissen ist von Umwelteinflüssen geprägt, da Honig von Bienen produziert wird, die Nektar und Pollen aus ihrer unmittelbaren Umgebung sammeln. Um das Metabolom biologischer Proben zu analysieren, ist das analytische Verfahren der Flüssigchromatographie mit Massenspektrometrie-Kopplung (LCMS) aufgrund ihres hohen Durchsatzes, der optionalen weichen Ionisierung und der breiten Erfassung bestimmter Verbindungsklassen sehr gut geeignet. Das Ziel dieses Projektes ist es, die Authentizitätanalyse von Honig mit einer geeigneten bioinformatischen Analyse der LCMS Daten zu ermöglichen.
Dieses Projekt ist in drei Teile gegliedert. Im ersten Teil wird ein Workflow entwickelt, durch den LCMS-Daten von verschiedenen Geräten genutzt werden können. Anschließend werden chemometrische Methoden wie die Hauptkomponentenanalyse (PCA), Soft Independent Modelling of Class Analogy (SIMCA) und Methoden des maschinellen Lernens, z. B. Random Forest (RF), zur Unterscheidung von Honigproben angewendet. Neben der Anwendung von RFs zur Klassifizierung werden RF-basierte Verfahren dabei auch zur Charakterisierung des Honigs verwendet. Dies wird dadurch erreicht, dass relevante Variablen selektiert werden, welche für die Unterschiede zwischen den Proben relevant sind und damit für die Interpretation spezifischer Umwelteinflüsse verwendet werden können. Darüber hinaus werden die Variablenbeziehungen der komplexen Daten mit Surrogate Minimal Depth (SMD) untersucht. SMD ermöglicht dabei die Identifizierung von gemeinsam vorkommenden Metaboliten und metabolischen Pathways, die mit bestimmten Umwelteinflüssen und somit mit der geografischen Herkunft des Honigs in Verbindung gebracht werden können.
Im zweiten Teil dieses Projekts wird die Verfälschung von Honig durch Zuckersirupe analysiert. Dazu werden multivariate Regressionsmodelle, z.B. durch Partial Least Squares (PLS) Regression oder RF erstellt, um den Anteil des dem Honig zugesetzten Sirups zu bestimmen. Auch hier werden Variablenselektionsverfahren eingesetzt, um spezifische Marker für Zuckersirupe zu identifizieren.
Die in den ersten beiden Teilen des Projekts entwickelten Methoden werden anschließend im dritten Teil des Projekts auf andere Bereiche der Lebensmittelauthentifizierung, wie z. B. die Verfälschung von Fruchtsäften, angewendet.
Dieses Projekt wurde von Jule Hansen bearbeitet.
Charakterisierung von Lebensmitteln anhand ihres Metaboloms
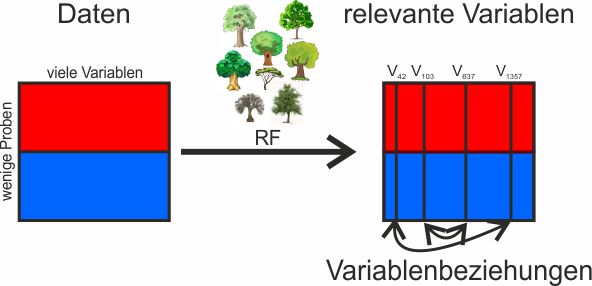
Die umfassende Analyse des Metaboloms wird für die Charakterisierung und Klassifizierung verschiedener biologischer Proben, einschließlich Lebensmitteln, immer wichtiger. Das Metabolom umfasst dabei mehrere sehr unterschiedliche Molekülklassen, weshalb verschiedene Analysetechniken für die Metabolomanalyse entwickelt wurden. Die meisten von ihnen basieren auf NMR und Massenspektrometrie. Bei der Lebensmittelanalyse wird Metabolomics zur Aufdeckung von Betrug eingesetzt, z.B. durch die Bestimmung der geografischen Herkunft und taxonomischer Merkmale.
In diesem Projekt werden bioinformatische Verfahren für die umfassende Analyse verschiedener Metabolomics-Daten und damit für die Klassifizierung, Charakterisierung und Authentifizierung von Lebensmitteln entwickelt. Dies wird durch die Anwendung etablierter Methoden wie der Hauptkomponentenanalyse (PCA) und Methoden des maschinellen Lernens, z. B. Random Forest (RF), erreicht. Letztere bieten verschiedene Vorteile für die Analyse von Metabolomics-Daten: Neben der Anwendung von RF zur Klassifizierung werden RF-basierte Ansätze auch zur Charakterisierung der untersuchten Lebensmittelproben eingesetzt. Dies geschieht durch die Selektion relevanter Variablen, die zur Interpretation von Unterschieden zwischen den Klassen und damit von spezifischen Einflüssen auf das Metabolom von Lebensmitteln verwendet werden. In diesem Zusammenhang werden verschiedene Selektionsverfahren angewendet und verglichen. Darüber hinaus werden die komplexen Eigenschaften des Metaboloms durch die Analyse von Variablenbeziehungen erfasst. Diese wird durch die Anwendung von Surrogate Minimal Depth (SMD) erreicht und ermöglicht die Identifizierung von gemeinsam vorkommenden Metaboliten, bzw. metabolischer Pathways, die mit spezifischen Umwelteinflüssen von Lebensmitteln in Verbindung gebracht werden können. Um dieses Wissen über funktionelle Beziehungen anschließend direkt in den Modellierungsprozess einzubeziehen, werden in diesem Projekt auch sog. pathway-guided RF Methoden angewendet.
Dieses Projekt wurde von Sören Wenck bearbeitet.
Analyse von Schriftartefakten
Dieses Projekt war Teil des Exzellenzclusters "Understanding Written Artefacts" und wurde von Lucas Voges bearbeitet. Weitere Informationen zum Projekt sind hier zu finden.
