Forschungsschwerpunkte
Molecular Modelling und Medizinische Chemie (Life Science)Das Verständnis von molekularen Mechanismen der Protein-Ligand-Wechselwirkungen ist eine Grundlage der Arzneistoffentwicklung. Auf dieser Basis können Struktur-Wirkungs-Beziehungen und Resistenzmechanismen auf molekularer Ebene erkannt und beim Design neuer Wirkstoffe berücksichtigt werden. In diesem Feld sind die verschiedenen Projekte angesiedelt. Um molekulare Wechselwirkungen der Inhibitoren von plasmodialer Dihydrofolat-Reduktase (DHFR) mit dem Zielenzym zu analysieren, wurden die Informationen aus Röntgenstrukturdaten von DHFR-Strukturen verschiedener Spezies genutzt und ein Homologie-Modell des Enzyms erstellt (Abbildung 1).  Das Modell erlaubt ein detailliertes Studium der Protein-Ligand-Wechselwirkungen und zeigt Möglichkeiten auf, beim Design neuer Wirkstoffe gegen Malaria molekulare Ansatzpunkte für Resistenzentwicklungen zu vermeiden (Lemcke, T., Christensen, I. T., Joergensen, F. S. (1999). Towards an understanding of drug resistance in Malaria: Three-dimensional structure of Plasmodium falciparum dihydrofolate reductase by homology building. Bioorganic & Medicinal Chemistry 7, 1003-1011). Die durch Punktmutationen im plasmodialen Enzym verursachten Resistenzen konnten mit Hilfe des Modells erklärt und verstanden werden (Modelling Studie im Arbeitskreis von Flemming Steen Joergensen, Universität Copenhagen/Dänemark). Die Enzymklasse der Kinasen erfüllt bei der Signal-Weiterleitung in der Zelle und bei der Zellteilung eine wichtige Rolle. Eine Vielzahl von Stoffwechselvorgängen wird von Kinasen beeinflusst oder kontrolliert. Bei einigen Erkrankungen sind die Kinasen zum Teil überexprimiert (z.B. verschiedene Tumorerkrankungen) oder hyperaktiv (Alzheimersche Krankheit). Selektive Kinase-Hemmstoffe sind aus diesem Grund sehr attraktive Zielstrukturen in der Wirkstoffentwicklung bei verschiedenen Krankheitbildern (Krebs, Diabetes, Alzheimer, etc.) Bei der Suche nach selektiven Inhibitoren verschiedener Kinasen (z.B. CDK1, CDK5, GSK-3b, c-MET) werden Methoden des Molecular Modelling wie Docking (Abbildung 2), 3D-QSAR und virtuelles Screening eingesetzt. So wurden für die Paullone, 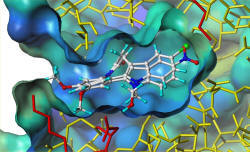 eine Gruppe neuer Kinase-Inhibitoren, durch Comparative Molecular Similarity Index-Analysen (CoMSIA) für CDK1, CDK5 und GSK-3b quantitative Struktur-Wirkungsbeziehungen errechnet (Kunick, C., Lauenroth, K., Wieking, K., Xie, X., Schultz, C., Gussio, R., Zaharevitz, D., Leost, M., Meijer, L., Weber, A., Joergensen, F. S. and Lemcke, T.*; Evaluation and Comparison of 3D-QSAR CoMSIA Models for CDK1, CDK5, and GSK-3 Inhibition by Paullones. J. Med. Chem. 2004, 47, 22-36. Daraus abgeleitete CoMSIA-Felder lassen sich bezüglich der molekularen Eigenschaften von neuen Inhibitoren interpretieren, wodurch die Vorhersage der Aktivität potentieller Hemmstoffe ermöglicht wird. In Kooperation mit den Gruppe von Matthias Rarey (ZBH) und Conrad Kunick (Pharmazie, Uni-Braunschweig) wurde ein virtuelles Screening zur Auffindung von selektiven Inhibitoren der c-Met Kinase durchgeführt. Die dabei gefundenen Hits werden mit Molecular Modelling und synthetischen Experimenten weiter untersucht und auf diese Weise neue Leitstrukturen für die Hemmung der c-Met Kinase entwickelt. Bei der Suche nach neuen Arzneistoffen zur Bekämpfung von Malaria ist die plasmodiale GSK-3 als potentielle Zielstruktur neuer Antimalariamittel identifiziert worden. Zum Design selektiver Inhibitoren dieser Kinase wurde zunächst ein Homologie-Modell, basierend auf der Struktur der homologen humanen GSK-3b erstellt (Kruggel, S.; Lemcke, T.; Generation and Evaluation of a Homology Model of PfGSK-3. Arch. Pharm. Chem. Life Sci. 2009, 342, 327 – 332) Mit Hilfe dieses Modells findet ein strukturbasiertes Design statt, dass die Aktivität und Selektivität vorhandener Inhibitoren verbessern soll. Mit Hilfe von molekularen Interaktionsfeldern (MIFás) und Dockingexperimenten wurden Vorschläge zu Bindungsmodus in der ATP-Bindetasche des Enzyms erhalten und weiterentwickelt. |
